2019年5月16日,3522vip浦京集团高琳团队联合费城儿童医院Tan Kai在Nature Communications上发表题为“Optimal control nodes in disease-perturbed networks astargets for combination therapy”的文章,描述了最优控制节点(OptiCon)算法,这是一个基因调控网络中协同关键调节因子的系统和从头识别的通用框架。
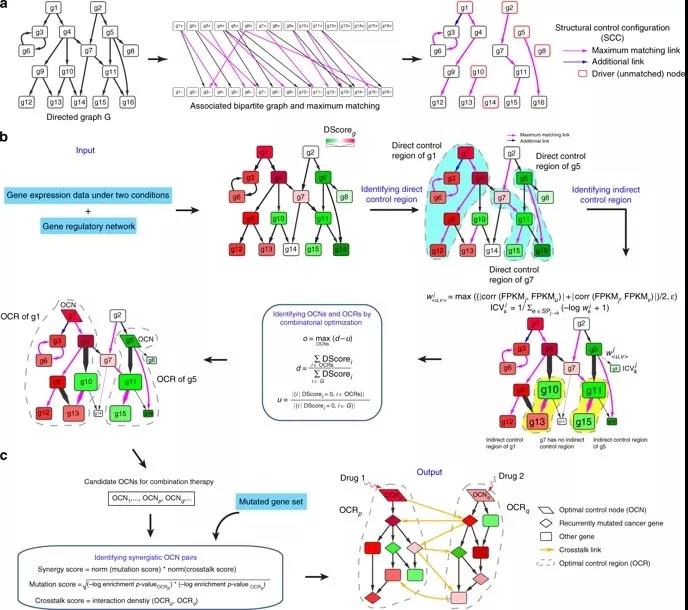
通过使用基因表达作为标准网络可控性框架中的约束,OptiCon首先在疾病扰动的基因调控网络中识别出一组最佳控制节点(OCN)。鉴定的OCN对失调的途径施加最大控制,但对未受疾病干扰的途径的控制最小。接下来,使用结合基因突变和基因功能相互作用信息的协同作用评分,OptiCon将一组协同OCN识别为疾病扰动网络中的关键调节因子,其可作为联合治疗的候选靶标。
研究人员开发OptiCon(最佳控制节点)算法,用于识别一组OCN,这些OCN最大限度地控制基因网络的解除管制部分,并最小化对网络未受干扰部分的控制。从网络中的所有基因搜索该组OCN,并且可以将其视为候选药物靶标。
首先通过组合来自三个手动策划的途径数据库,Reactome,京都基因和基因组百科全书(KEGG)和NCI-自然途径相互作用数据库的条目,构建了高质量的基因调控网络。然后,将网络与来自三种癌症类型,肝细胞癌(HCC),肺腺癌(LUAD)和乳腺浸润癌(BRCA)的基因表达数据组合,以构建三个解除管制的网络作为算法的输入。作为基线比较,将OptiCon与基于网络可控性理论的方法,TargetControl以及基于网络的度数和度外分布的方法进行了比较。与OptiCon类似,TargetControl识别一组节点,这些节点有效地控制网络中预先选定的一组其他节点。TargetControl和基于程度的方法不能预测协同调节剂。
因此,研究人员将OptiCon与另外两种预测协同调节因子的方法进行了比较,即富集调节分析(VIPER)和RACS9的蛋白质活性虚拟推断。VIPER使用主调节推断算法基于基因表达数据和基因调控网络鉴定协同主调节因子。RACS是一种半监督学习方法,其结合药物药理学特征,药物靶向网络和转录组学谱,以鉴定现有癌症药物的潜在协同组合。OptiCon分别为HCC,LUAD和BRCA确定了15,23和15个OCN。相比之下,TargetControl和VIPER通过TargetControl识别HCC,LUAD和BRCA中的更多数量的控制节点593,727和660,以及VIPER识别250,453和442。如此大量的控制节点代表了后续研究的艰巨挑战,特别是用于鉴定协同基因对。
大多数联合疗法是基于现有药物的目标开发的,现有药物仅代表人类蛋白质组的一小部分。研究人员引入了基于网络可控性的方法OptiCon,用于从头识别协同调节剂作为联合治疗的候选者。这些调节剂共同对失调的基因进行最大程度的控制,但对疾病中未受干扰的基因的控制最小。使用来自三种癌症类型的数据,显示68%的预测调节剂是已知的药物靶标或在癌症发展中具有关键作用。对于与副作用相关的已知蛋白质,预测的调节剂被耗尽。预测的协同作用由疾病特异性和临床相关的合成致死相互作用和实验验证支持。由协同调节剂调节的基因的很大一部分参与共同调节的子网络之间的密集相互作用并且有助于治疗抗性。 OptiCon代表了系统和从头识别细胞状态转变的协同调节因子的一般框架。
据悉,联合治疗通过对多种致病途径发挥作用,是对抗药物抗性和疾病异质性的有效策略。自从儿童急性淋巴细胞白血病联合化疗取得首次成功以来,联合治疗已被开发用于治疗许多复杂疾病,包括癌症,传染病和神经退行性疾病。近年来,已经开发了许多用于发现组合治疗靶标的计算方法,包括基于网络的方法。这些基于网络的方法的动机是观察到多种疾病相关基因而不是单一基因经常在复杂基因网络内相互作用,导致疾病表型和耐药性。基于网络的代表性方法是抗癌协同(RACS)算法的排序系统。它预测使用药物处理的转录组谱和蛋白质 - 蛋白质相互作用网络和KEGG途径的协同抗癌药物。Drug Combo Ranker是使用药物处理的基因表达谱鉴定已知药物组合的另一种基于网络的代表性方法。大多数现有方法侧重于确定现有药物的协同组合。然而,现有药物的目标仅限于一小组蛋白质。截至2016年,仅有667种人类蛋白质作为批准药物的靶标。因此,大量基因及其组合仍未作为潜在的组合治疗靶标进行探索。
图文转自微信公众号:iNature
原文连接:https://www.nature.com/articles/s41467-019-10215-y



